Inledning
Inledning
 Restriktionsenzymer är en slags enzymer som klyver DNA. Detta kan man använda på olika sätt för att identifiera DNA eller klyva upp ett helt genom i mindre, hanterbara bitar. I den här laborationen ska du klippa upp DNA från bakteriofag λ, och sedan analysera det med hjälp av agarosgelelektrofores.
Restriktionsenzymer är en slags enzymer som klyver DNA. Detta kan man använda på olika sätt för att identifiera DNA eller klyva upp ett helt genom i mindre, hanterbara bitar. I den här laborationen ska du klippa upp DNA från bakteriofag λ, och sedan analysera det med hjälp av agarosgelelektrofores.
Tid för detta
Laborationen tar 2-3 laborationstillfällen i anspråk. Vid det första laborationstillfället ingår 20-30 minuters väntetid medan restriktionsenzymerna arbetar och agarosgelen får stelna. Denna tid kan man t.ex. använda till att förklara principen för agarosgelelektrofores. Vid det andra laborationstillfället ska proverna appliceras och gelen köras. Själva appliceringen bör inte ta mer än 20 minuter, men hur länge gelen skall köras beror på vilket elektroforessystem man använder. Även här blir det alltså väntetid, som man bör använda på något vettigt vis. Fotografering och avläsning av gelen är en relativt snabb process, som inte bjuder på några längre väntetider.
Material
Varje grupp behöver:
- Mikropipett, 1-10 μl
- Mikropipett, 50 μl
- Mikropipett, 200 μl
- Ett eppendorfrör med 10 μg λ-DNA löst i avjonat eller destillerat vatten.
- Tre eppendorfrör med 10 units av olika restriktionsenzymer i, t.ex. EcoRI, HindIII och BamHI. En unit är den mängd enzym, som klipper 1 μg DNA på en timme vid 37ºC. ”Unit” är inte någon väldefinierad enhet, utan varierar ofta från enzym till enzym och från tillverkare till tillverkare.
För att restriktionsenzymerna ska fungera optimalt, krävs det att de befinner sig i någon form av buffert. Ofta kan man köpa restriktionsenzymer som frystorkats tillsammans med sin buffert, så att man bara behöver tillsätta vatten till dem.
Tänk på att när man tillsätter DNA-lösningen till restriktionsenzymet späds bufferten. Bufferten måste därför vara så pass koncentrerad att slutkoncentrationen blir den riktiga först när man tillsatt sin DNA-lösning.
- Ett fjärde eppendorfrör utan restriktionsenzym för negativ kontroll.
- En flytplatta av frigolit
- Ett eppendorfrör med 10× laddningsfärg (bromfenolblå)
- Material till agarosgelelektrofores av DNA
- Storleksmarkör (DNA-stege)
- En linjal
Gemensamt behövs:
- Ett vattenbad på 37 ºC.
Gör såhär
Klyv ert DNA (dag 1)
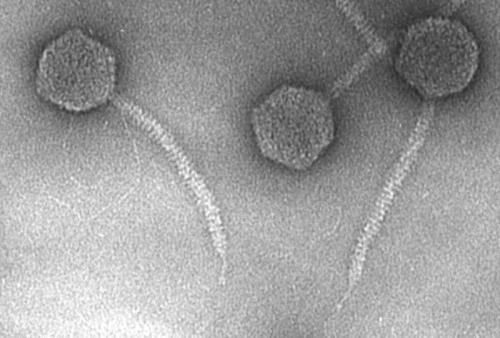
 Elektronmikroskopbild av bakteriofag λ.
Elektronmikroskopbild av bakteriofag λ.
- För över en volym, som motsvarar 2 μg DNA till vart och ett av rören med restriktionsenzymer, samt det tomma eppendorfröret (kontrollröret). Använd en ny pipettspets till varje rör.
Om vätskan hamnar i en liten droppe på kanten, kan man centrifugera ner den i en liten bordscentrifug eller mikrocentrifug. Ännu enklare är att svänga kraftigt med armen (à la Gunde Svan eller Margareta Forsberg), så att vätskan åker ner p.g.a. centrifugalkraften.
- Märk alla fyra eppendorfrören, och tryck ner dem genom flytplattan så att i alla fall spetsen sticker ut undertill. Inkubera sedan hela paketet i vattenbadet på 37 ºC i 30-45 minuter. Knäpp på rören 2-3 gånger under inkuberingen, för att se till att allt hålls väl blandat i rören.
- Medan DNA:t klyvs, kan man passa på att gjuta gelen.
- Tillsätt laddningsfärg till vart och ett av de fyra eppendorfrören med DNA i. Laddningsfärgen skall spädas 1 + 9, så man skall tillsätta 1/9 av volymen som redan är i röret.
- Det kluvna DNA:t kan också sparas i kylskåp till nästa laborationstillfälle.
Kör er gel (dag 2)
- Applicera proverna på gelen och kör den enligt tillverkaren rekommendationer eller de här instruktionerna.
- När laddningsfärgen nått kanten av gelen, kan elektroforesen avbrytas. Hur lång tid detta tar varierar kraftigt, beroende på vilken typ av elektroforeskärl man använder.
- Lägg den färdigkörda gelen på ett UV-bord och fotografera av den.
Läs av er gel (dag 3)
|
Restriktionsenzym |
Klyver vid baspar nr. |
Antal bildade fragment |
|
BamHI |
5 505 22 346 27 972 34 499 41 732 |
6 st. |
|
EcoRI |
21 226 26 104 31 747 39 168 44 972 |
6 st. |
|
HindIII |
23 130 25 157 27 479 36 895 37 459 37 584 44 141 |
8 st. |
- Mät med linjal eller på datorskärmen hur många mm de olika stora fragmenten vandrat i gelen.
- Mät hur många mm fragmenten från varje klyvningsreaktion (eppendorfrör) vandrat.
- Jämför storleken på fragmenten från varje klvyningsreaktion med storleksmarkören.
- Jämför storleken på fragmenten från de olika klyvningsreaktionerna.
Frågor att besvara
- Sätt av i ett diagram vandringssträckan mot logaritmen av storleken på fragmenten från storleksmarkören.
- Sätt in vandringssträckan från de bildade DNA-fragmenten för de olika klyvningsreaktionerna i samma diagram
- Hur många baspar består fag λ:s DNA-molekyl av?
- Ta reda på vid vilka baspar i DNA-molekylen från fag λ som de olika restriktionsenzymerna klipper (denna information tillhandahålls ofta av tillverkaren av restriktionsenzymerna eller av fag λ-DNA:t; se också tabellen intill). Stämmer era resultat överens med detta? Om inte, vad tror ni det beror på?
Riskanalys
Laborationen är inte riskfylld. Observera dock riskanalysen som hänger samman med hur man analyserar sitt DNA med agarosgelelektrofores!
Tillkännagivande
Den här laborationen har jag inte kommit på själv, utan inspirerats av från NCBE. Jag har dock skrivit om den och anpassat den till mina behov.